La dynamique moléculaire pour contrer les infections
Département de biochimie, microbiologie et bio-informatique, Université Laval
Mode d’action de la protéine de l’hémoglobine tronquée dans le processus d’infection de la bactérie de la tuberculose

Patrick Lagüe, biophysicien
La tuberculose infecte le tiers de la population mondiale. Chez la majorité des gens infectés, le système immunitaire réussit à arrêter la progression de la maladie en produisant le gaz NO et en diminuant la concentration d’oxygène au niveau de la lésion infectée. Dans ces conditions, le NO produit un effet toxique qui affecte la respiration de la bactérie et la maintient dans un état de latence sans conséquences pour l’organisme. Mais la bactérie de tuberculose a plus d’un tour dans son sac pour déjouer le système immunitaire. L’un  de ces mécanismes consiste en la synthèse de l’hémoglobine tronquée N (trHbN). Cette protéine réussie à protéger la respiration de la bactérie de l’effet toxique du NO en transformant celui-ci en nitrate. En raison de son rôle dans la survie de la bactérie de tuberculose, la caractérisation structurale et fonctionnelle de trHbN est devenue un sujet d’intérêt. Pour ce faire on utilise des modèles de dynamique moléculaire qui consistent à simuler le mouvement d’un groupe d’atomes (6000) dans le temps pour des conditions connues. Un algorithme de dynamique est utilisé pour le calcul des forces ainsi que les vitesses et positions des atomes qui informent sur la trajectoire de la molécule pendant un intervalle de temps spécifique (17.9 µs). De tels calculs demandent 150 cœurs-année du superordinateur nommé Colosse. Ces simulations ont permit d’identifier l’aspect particulier de la protéine qui lui procure sa réactivité face au NO. Des cavités se trouvent dans la structure très rigide de la protéine. La nature hydrophobique de ces cavités force les molécules d’eau à s’éloigné de la surface de la protéine ce qui favorise la capture de NO. Pour mieux comprendre l’impact du milieu sur la structure et la fonction de la protéine, les modèles de dynamique moléculaire ont aussi servis à l’étude de la protéine dans un milieu de différentes compositions. La magnitude de l’interaction de la protéine avec une membrane biologique dépend de la nature des lipides dont elle est composée. L’interaction est plus grande avec le lipide cardiolopin qui se retrouve presqu’exclusivement dans les membranes qui construisent les murs de la bactérie. Cet environnement avantage l’activité enzymatique de la protéine.
de ces mécanismes consiste en la synthèse de l’hémoglobine tronquée N (trHbN). Cette protéine réussie à protéger la respiration de la bactérie de l’effet toxique du NO en transformant celui-ci en nitrate. En raison de son rôle dans la survie de la bactérie de tuberculose, la caractérisation structurale et fonctionnelle de trHbN est devenue un sujet d’intérêt. Pour ce faire on utilise des modèles de dynamique moléculaire qui consistent à simuler le mouvement d’un groupe d’atomes (6000) dans le temps pour des conditions connues. Un algorithme de dynamique est utilisé pour le calcul des forces ainsi que les vitesses et positions des atomes qui informent sur la trajectoire de la molécule pendant un intervalle de temps spécifique (17.9 µs). De tels calculs demandent 150 cœurs-année du superordinateur nommé Colosse. Ces simulations ont permit d’identifier l’aspect particulier de la protéine qui lui procure sa réactivité face au NO. Des cavités se trouvent dans la structure très rigide de la protéine. La nature hydrophobique de ces cavités force les molécules d’eau à s’éloigné de la surface de la protéine ce qui favorise la capture de NO. Pour mieux comprendre l’impact du milieu sur la structure et la fonction de la protéine, les modèles de dynamique moléculaire ont aussi servis à l’étude de la protéine dans un milieu de différentes compositions. La magnitude de l’interaction de la protéine avec une membrane biologique dépend de la nature des lipides dont elle est composée. L’interaction est plus grande avec le lipide cardiolopin qui se retrouve presqu’exclusivement dans les membranes qui construisent les murs de la bactérie. Cet environnement avantage l’activité enzymatique de la protéine.
Mode d’action du peptide de fusion dans le processus d’infection du virus de l’influenza
Lors de l’infection du virus de l’influenza, celui-ci pénètre dans les cellules de l’organisme puis s’intègre à la membrane de l’endosome. 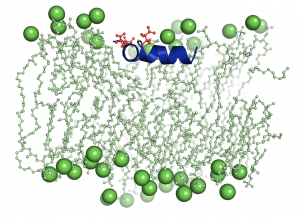 L’endosome est un compartiment dans la cellule qui trie les protéines, recyclant celles qui se retrouvent dans sa membrane et dégradant les autres. Le PH basique de l’endosome induit l’insertion dans sa membrane de la protéine viral hemagglutinin et donc son recyclage. C’est plus particulièrement un de ses segments de la protéine, qu’on nomme peptide de fusion, qui vient en contact avec la membrane. La compréhension de ce phénomène de fusion est essentielle au développement thérapeutique pour enrayer l’infection. Des simulations de dynamique moléculaire d’un groupe de 30000 atomes pendant un intervalle de 10 µs sont utilisées pour étudier le comportement de la structure moléculaire du peptide de fusion en présence de membranes lipidiques selon le PH. De tels calculs demandent 44 cœurs-année du superordinateur Colosse.
L’endosome est un compartiment dans la cellule qui trie les protéines, recyclant celles qui se retrouvent dans sa membrane et dégradant les autres. Le PH basique de l’endosome induit l’insertion dans sa membrane de la protéine viral hemagglutinin et donc son recyclage. C’est plus particulièrement un de ses segments de la protéine, qu’on nomme peptide de fusion, qui vient en contact avec la membrane. La compréhension de ce phénomène de fusion est essentielle au développement thérapeutique pour enrayer l’infection. Des simulations de dynamique moléculaire d’un groupe de 30000 atomes pendant un intervalle de 10 µs sont utilisées pour étudier le comportement de la structure moléculaire du peptide de fusion en présence de membranes lipidiques selon le PH. De tels calculs demandent 44 cœurs-année du superordinateur Colosse.
Design rationnel de nouveaux antibiotiques
Propriétés dynamiques des β-lactamases
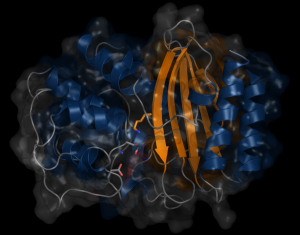 Les beta-lactamines sont les antibiotiques les plus utilisés sur le marché. Les enzymes beta-lactamases qu’on retrouvent dans certaines bactéries sont capable de couper les molécules beta-lactam ce qui confère à ces bactéries une résistance à ce type d’antibiotique. Ainsi, par exemple, l’enzyme TEM-1 rend la bactérie E. coli résistante à l’ampicillin, tandis que PSE-4 rend la bactérie P. aeruginosa résistante à la carbenicillin. Afin de faciliter l’ingénierie de nouveaux antibiotiques, il est important d’acquérir plus d’information sur ce mécanisme de résistance bactérienne. On doit comprendre les variations structurales des molécules TEM-1 et PSE-4 qui permet la reconnaissance et la catalyse des antibiotiques beta-lactamines. Les propriétés dynamiques de ces β-lactamases seront donc caractérisées grâce à une approche combinant simulations par dynamique moléculaire et spectroscopie RMN qui mesure les interactions magnétiques entre les molécules.
Les beta-lactamines sont les antibiotiques les plus utilisés sur le marché. Les enzymes beta-lactamases qu’on retrouvent dans certaines bactéries sont capable de couper les molécules beta-lactam ce qui confère à ces bactéries une résistance à ce type d’antibiotique. Ainsi, par exemple, l’enzyme TEM-1 rend la bactérie E. coli résistante à l’ampicillin, tandis que PSE-4 rend la bactérie P. aeruginosa résistante à la carbenicillin. Afin de faciliter l’ingénierie de nouveaux antibiotiques, il est important d’acquérir plus d’information sur ce mécanisme de résistance bactérienne. On doit comprendre les variations structurales des molécules TEM-1 et PSE-4 qui permet la reconnaissance et la catalyse des antibiotiques beta-lactamines. Les propriétés dynamiques de ces β-lactamases seront donc caractérisées grâce à une approche combinant simulations par dynamique moléculaire et spectroscopie RMN qui mesure les interactions magnétiques entre les molécules.
La Glutamyl-tRNA synthetase comme cible thérapeutique
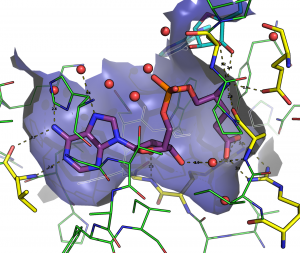 La Glutamyl-tRNA synthetase est une protéine bactérienne essentielle. L’inhibition de cette protéine entraîne éventuellement sa mort. Ainsi, avec pour but le développement d’une nouvelle série d’antibiotique, on étudie les inhibiteurs de la GluRS et plus précisément les interactions entre les molécules de la protéine et 50 différents ligands. Pour caractériser ces interactions, les modèles de dynamique moléculaire se composent de 60000 atomes dans un intervalle de temps de 25µs ce qui requière 100 cœur-année sur Colosse. Deux autres techniques sont utilisées, soit le docking(fitted) et la mécanique quantique(GAMESS).
La Glutamyl-tRNA synthetase est une protéine bactérienne essentielle. L’inhibition de cette protéine entraîne éventuellement sa mort. Ainsi, avec pour but le développement d’une nouvelle série d’antibiotique, on étudie les inhibiteurs de la GluRS et plus précisément les interactions entre les molécules de la protéine et 50 différents ligands. Pour caractériser ces interactions, les modèles de dynamique moléculaire se composent de 60000 atomes dans un intervalle de temps de 25µs ce qui requière 100 cœur-année sur Colosse. Deux autres techniques sont utilisées, soit le docking(fitted) et la mécanique quantique(GAMESS).


